复现一篇生信文章有多难,到处都是坑。。。
今天本来打算复现一篇去年发表在 OncoTargets and Therapy GEO数据库整合挖掘的论文,这个杂志素来以灌输闻名,“水的”已经不是SCI了,但是影响因子还是3左右,按理说文章质量应该尚可,而且这又是一篇纯数据挖掘的文章,所以我就去打开了我的Rstudio,开始数据分析,在做到差异表达分析的时候被作者的操作惊到了,到底怎么回事呢,首先我们看看文献中怎么说的,作者首先分析了GDS35
今天本来打算复现一篇去年发表在 OncoTargets and Therapy 上关于GEO数据库整合挖掘的论文,这个杂志素来以灌水闻名,“水的”已经不是SCI了,但是影响因子还是3左右,按理说文章质量应该尚可,而且这又是一篇纯数据挖掘的文章,所以我就去打开了我的Rstudio,开始数据分析,在做到差异表达分析的时候被作者的操作惊到了,到底怎么回事呢,首先我们看看文献中怎么说的,作者首先分析了GDS3592数据集,这是差异表达结果: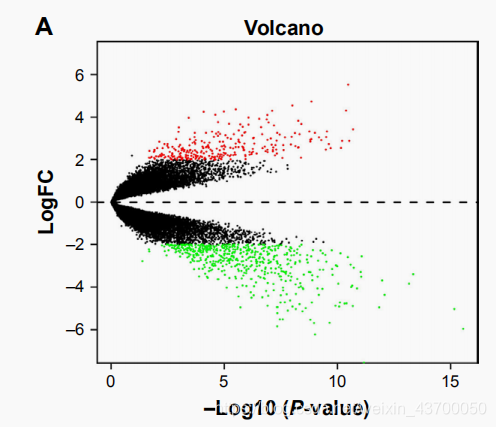

上面三张图总结一下就是:
- logFC >2 并且correccted P-value < 0.05为上调基因,共550个,用红色点表示
- logFC < -2 并且correccted P-value < 0.05下调基因,共289个,用绿色点表示
这时候如果你仔细看第一张图,你可以明显的感觉到,明明绿色点比红色点多啊,这不是与作者结果相反吗?难道作者还会犯这么低级的错误嘛,可是我也不是红绿色盲啊,本着科学的精神,我就对作者的分析进行了复现。
下面是我复现结果:
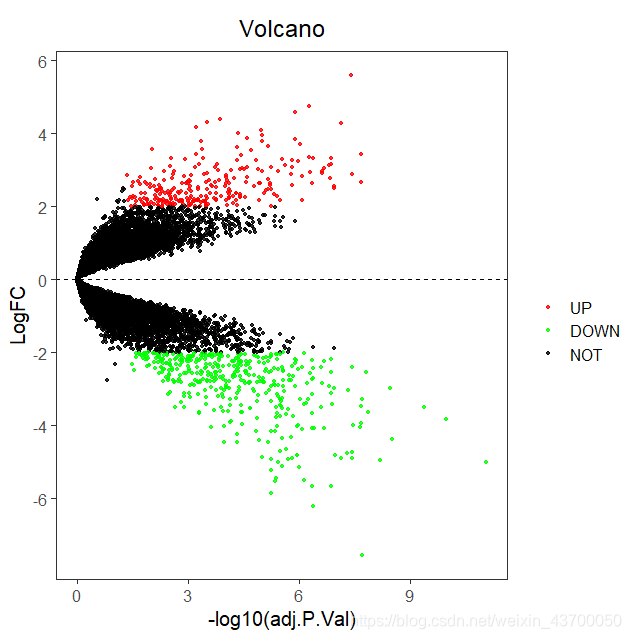
经过对比发现有以下不同:在使用矫正P值作为筛选条件时,1、2两处的差异基因明显与作者原图中的点不同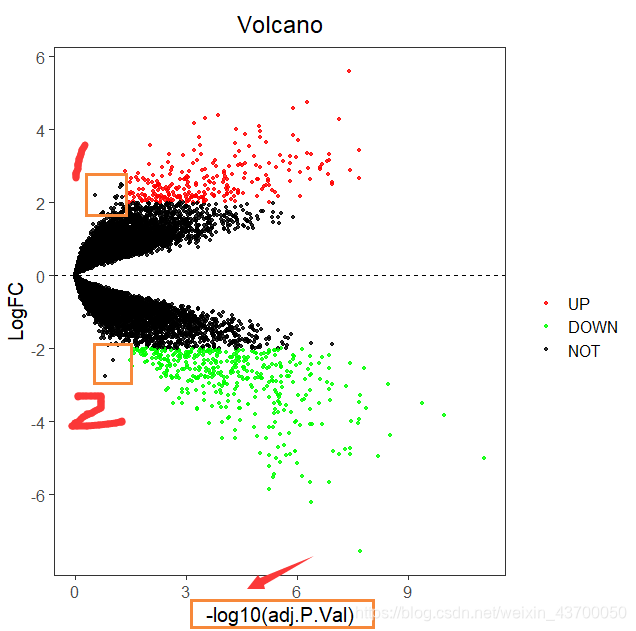
于是我便直接用P值作为筛选条件,得到以下图形: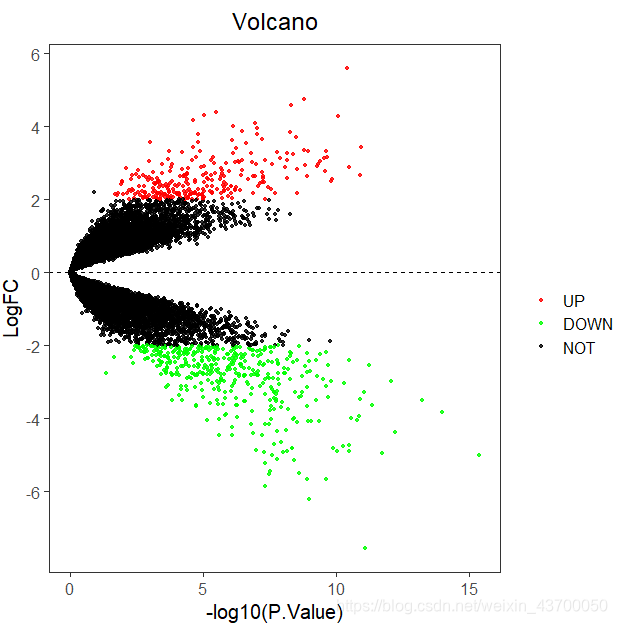
哇!终于和原图几乎一模一样了,可是问题来了,作者明明说用的correct P-value作为筛选,经过绘图发现,使用的却是P-value筛选,这两种筛选条件所选择的差异表达基因是有所不同的,这是遇到的另外一个问题!
**
接下来就是我们一开始遇到的问题了
**
这是我差异表达基因的结果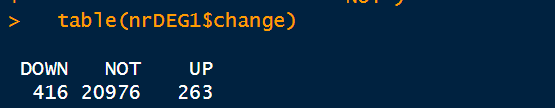
上调基因263,下调基因416,下调基因明显多于上调基因,这种结果与图片上的点的分布是一致的,所以,我想结论出来了:
- 作者把差异表达基因写反了,写反了,写反了!!!
- 作者也没有使用correct P-value < 0.05 作为筛选差异表达的标准,而是直接使用的P-value < 0.05,这样会增加假阳性结果,明明不是差异表达基因,却被筛选为差异表达基因。
- 这才是开始就踩了两个坑,文章复现真的好难啊!
本博客内容将同步更新到个人微信公众号:生信玩家。欢迎大家关注~~~
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容






所有评论(0)